Google ColaboratoryでPyStanをいじくる
ベイズモデリングのお勉強で「StanとRでなんちゃらhogehoge」的な書籍のサンプルコードを実装しようとしているのですが、ここはRではなく使い慣れているPythonとGoogle Colabで動かしたい。と思ってググってみたところ、ローカルで動かそうと思うと意外と面倒なStanですが、幸運なことにGoogle Colabでは既にインストール済みらしく、importして即使えるようです。
ってことで、Google ColabでGLMのパラメータをMCMCで推定する流れを備忘録として残しておきます。
2022/08/28 追記 importについて情報更新があります。以下をご参照ください。
実装
以下のページを参考にさせていただきました(というか、チュートリアルとしてほぼ同じことトレースさせていただきました)。
1. ライブラリのインポート
必要なライブラリをインポートしていきます。っていうかこれら全部インストール済みで、pipとかで改めてインストールする必要がないのはマジ有難い。
#pystanのインポート import pystan #作図用ライブラリ import matplotlib.pyplot as plt import seaborn as sns %matplotlib inline #MCMCの結果可視化用ライブラリ import arviz #いつもの import pandas as pd import numpy as np
2. データの読み込み
モデリングの対象となるデータセットを読み込んでpandasのデータフレームに変換します。
なお、データはこちらで手に入るAuto MPGデータセットを利用します。 MPG=Miles Per Gallonで1ガロンあたりの走行距離、つまり自動車の燃費のデータですね。
work_path = "データの置き場所/" # データの読み込み column_names = ['MPG','Cylinders','Displacement','Horsepower','Weight', 'Acceleration','Model Year','Origin'] df = pd.read_csv(work_path + "auto-mpg.data", names=column_names, na_values = "no_data", comment='\t', sep=" ", skipinitialspace=True) df
| MPG | Cylinders | Displacement | Horsepower | Weight | Acceleration | Model Year | Origin | |
|---|---|---|---|---|---|---|---|---|
| 0 | 18.0 | 8 | 307.0 | 130.0 | 3504.0 | 12.0 | 70 | 1 |
| 1 | 15.0 | 8 | 350.0 | 165.0 | 3693.0 | 11.5 | 70 | 1 |
| 2 | 18.0 | 8 | 318.0 | 150.0 | 3436.0 | 11.0 | 70 | 1 |
| 3 | 16.0 | 8 | 304.0 | 150.0 | 3433.0 | 12.0 | 70 | 1 |
| 4 | 17.0 | 8 | 302.0 | 140.0 | 3449.0 | 10.5 | 70 | 1 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 393 | 27.0 | 4 | 140.0 | 86.00 | 2790.0 | 15.6 | 82 | 1 |
| 394 | 44.0 | 4 | 97.0 | 52.00 | 2130.0 | 24.6 | 82 | 2 |
| 395 | 32.0 | 4 | 135.0 | 84.00 | 2295.0 | 11.6 | 82 | 1 |
| 396 | 28.0 | 4 | 120.0 | 79.00 | 2625.0 | 18.6 | 82 | 1 |
| 397 | 31.0 | 4 | 119.0 | 82.00 | 2720.0 | 19.4 | 82 | 1 |
3. パラメータ推定
今回は目的変数の燃費(MPG)を正規分布でフィッティングしていきます。ここで、正規分布の平均は、線形予測子を(やや唐突ではありますが、先述の参考リンク先の情報に則って、、、)重量の逆数(1/weight)を説明変数とする線形関数とします。リンク関数は恒等関数です。
3.1. データを可視化
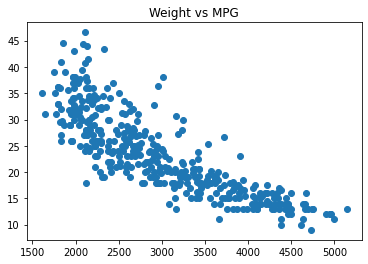
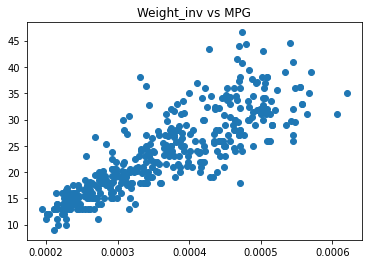
なにはともあれ、まずはデータの分布を見ましょう。なるほど、燃費と重量の逆数は正の相関がありそうですね。
全然余談ですけど、僕なら燃費と重量のプロットをみた瞬間に「負の相関だなぁ」と思ってそのままモデリングしちゃうと思うので、この分布を見て「逆数なら燃費と綺麗に相関するのでは?」との考えに至るの、すごい。
# 一応データ処理用のDFにコピってから後の処理にまわす df_prcd = df.copy() # 重さと燃費の関係 plt.scatter(df_prcd['Weight'], df_prcd['MPG']) plt.title("Weight vs MPG") plt.show() # 1/重さ と燃費の関係 df_prcd['Weight_inv'] = df_prcd['Weight'].apply(lambda x: 1/x) plt.scatter(df_prcd['Weight_inv'], df_prcd['MPG']) plt.title("Weight_inv vs MPG") plt.show()
3.2. モデルのコンパイル
書籍等を読む限りでは、Stanの利用方法としては、モデルとパラメータを記述した.stan拡張子のファイルを用意してRまたはPythonのスクリプト中でこれを読み込んでコンパイルする、という流れが正攻法のようでした。が、Stanファイルに記載する相当の内容をPythonスクリプト中に記載してそのままコンパイルすることもできるみたいなので、今回はこの方法で記述します(というか、これで書けるならわざわざStanファイルを用意する理由がわからなかった、、、)。
ちなみに、コンパイルに意外と時間かかるなぁと思って計測してみたら、ざっくり1分半くらいかかってるみたいですね。
%%time stan_model = """ data { int N; real X[N]; real Y[N]; } parameters { real a; real b; real<lower=0> sigma; } model { for (n in 1:N) { Y[n] ~ normal(a * X[n] + b, sigma); } } """ sm = pystan.StanModel(model_code=stan_model)
INFO:pystan:COMPILING THE C++ CODE FOR MODEL anon_model_baea8ca869534afad1a31f3e70798325 NOW. CPU times: user 2.17 s, sys: 160 ms, total: 2.33 s Wall time: 1min 27s
3.3. パラメータ推定
さて、コンパイルが終わったら、いざいざMCMCを回してパラメータを推定していきます。そこそこ時間がかかることを覚悟していましたが、10秒くらいであっさり終わってしまいました。探索するパラメータもデータ量も少ないからですかね。
出てきた結果を見ると、Rhatが1.0ということで収束は問題なさそう。
%%time
stan_data = {
'N': df_prcd.shape[0],
'X': df_prcd['Weight_inv'],
'Y': df_prcd['MPG']
}
fit = sm.sampling(data=stan_data, iter=2000, warmup=500, chains=4, seed=123)
print(fit)
Inference for Stan model: anon_model_baea8ca869534afad1a31f3e70798325. 4 chains, each with iter=2000; warmup=500; thin=1; post-warmup draws per chain=1500, total post-warmup draws=6000. mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat a 6.6e4 49.07 2130.6 6.2e4 6.5e4 6.6e4 6.8e4 7.0e4 1885 1.0 b -0.55 0.02 0.8 -2.15 -1.09 -0.53 -0.02 1.02 1877 1.0 sigma 4.25 2.7e-3 0.15 3.96 4.15 4.24 4.35 4.57 3193 1.0 lp__ -772.7 0.03 1.22 -775.9 -773.3 -772.4 -771.8 -771.3 1805 1.0 Samples were drawn using NUTS at Sat May 21 18:23:26 2022. For each parameter, n_eff is a crude measure of effective sample size, and Rhat is the potential scale reduction factor on split chains (at convergence, Rhat=1). CPU times: user 116 ms, sys: 47.8 ms, total: 163 ms Wall time: 9.17 s
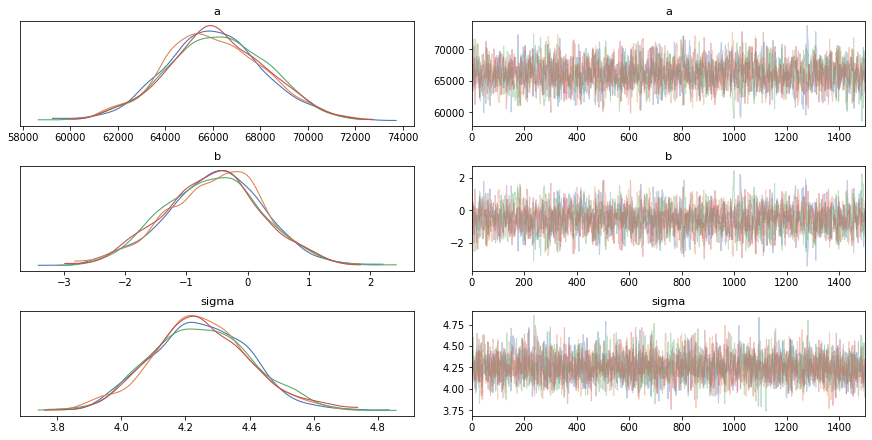
3.4. MCMCの結果の確認(トレースプロット)
トレースプロットを見ても、各チェーンが混ざっていて収束に関しては特に問題なさそうです。
fig = arviz.plot_trace(fit, compact=False, backend_kwargs={"constrained_layout":True})
ってことで、とりあえずパラメータ推定ができたのでGoogle Colabでの動作確認としてはここまでとします。
おわりに
はい。動作環境ができてしまったので、これでやらない言い訳はなくなりました。しばらくMCMCと仲良く戯れたいと思います。